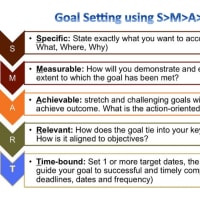
Rでhistgramは簡単に生成できるが、結構厄介な点もある。
まず基本的なhist()関数での生成を簡単に書いておく。
デモデーターを生成
> a <- rnorm(100,20,5)
> b <- rnorm(60, 10,3)
> c <- rnorm(80, 5,3)
> d <- rnorm(100,20,5)
> e <-rnorm(60,10,4)
> f <- rnorm(80,5,5)
まずはテストで
>hist(a)
と入力すると白黒のヒストグラムを簡単に作ってくれる。
これだと芸がないので、色をつけて、X軸のレンジとピッチサイズをbreaksで指定する。
>hist(a, col="blue", breaks=seq(-5,40,2.5))

同様にデーターb、cについても
>hist(b, col="red", breaks=seq(-5,40,2.5))
で、

>hist(c, col="green", breaks=seq(-5,40,2.5))
で

とここまでは簡単に生成できる。
次に重ね合わせ、二つ目以降の式でadd=Tとすればよいから、
>hist(a, col="blue", breaks=seq(-5,40,2.5))
>hist(b, col="red", breaks=seq(-5,40,2.5), add=T)
>hist(c, col="green", breaks=seq(-5,40,2.5), add=T)

Y軸のレンジをylim=c(1,40)で指定してちょい修正。
>hist(a, col="blue",ylim=c(1,40),breaks=seq(-5,40,2.5))
>hist(b, col="red", breaks=seq(-5,40,2.5), add=T)
>hist(c, col="green", breaks=seq(-5,40,2.5), add=T)
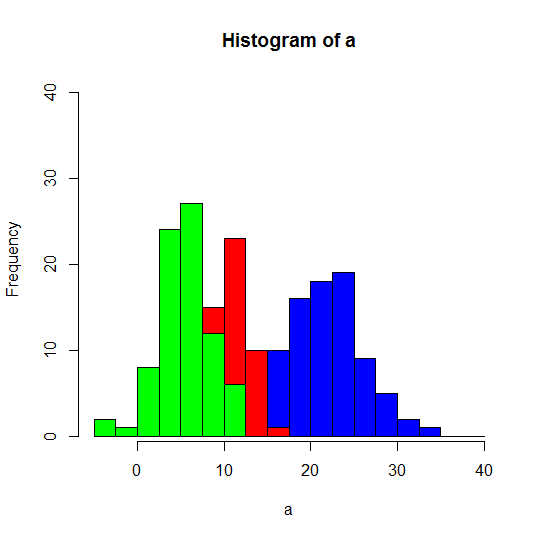
やはり重ね合わせはうまくいかない。
特に6つのグラフが重なるような状況になると悲惨である。
>hist(a, col="blue",ylim=c(1,40),breaks=seq(-15,40,2.5))
>hist(b, col="red", breaks=seq(-15,40,2.5), add=T)
>hist(c, col="green", breaks=seq(-15,40,2.5), add=T)
>hist(d, col="orange", breaks=seq(-15,40,2.5), add=T)
>hist(e, col="yellow", breaks=seq(-15,40,2.5), add=T)
>hist(f, col="purple", breaks=seq(-15,40,2.5), add=T)

とまあひどい感じの重ね合わせになる。
一応biostaticsのページにreshape2とggplot2をつかってやる綺麗にhistgramを重ね合わせる方法がでているのであるが、このページと同じ内容をおこなってもmelt()がうまく働いてくれない(*)
(*)なんとなくバグっぽい!!!
まず基本的なhist()関数での生成を簡単に書いておく。
デモデーターを生成
> a <- rnorm(100,20,5)
> b <- rnorm(60, 10,3)
> c <- rnorm(80, 5,3)
> d <- rnorm(100,20,5)
> e <-rnorm(60,10,4)
> f <- rnorm(80,5,5)
まずはテストで
>hist(a)
と入力すると白黒のヒストグラムを簡単に作ってくれる。
これだと芸がないので、色をつけて、X軸のレンジとピッチサイズをbreaksで指定する。
>hist(a, col="blue", breaks=seq(-5,40,2.5))

同様にデーターb、cについても
>hist(b, col="red", breaks=seq(-5,40,2.5))
で、

>hist(c, col="green", breaks=seq(-5,40,2.5))
で

とここまでは簡単に生成できる。
次に重ね合わせ、二つ目以降の式でadd=Tとすればよいから、
>hist(a, col="blue", breaks=seq(-5,40,2.5))
>hist(b, col="red", breaks=seq(-5,40,2.5), add=T)
>hist(c, col="green", breaks=seq(-5,40,2.5), add=T)

Y軸のレンジをylim=c(1,40)で指定してちょい修正。
>hist(a, col="blue",ylim=c(1,40),breaks=seq(-5,40,2.5))
>hist(b, col="red", breaks=seq(-5,40,2.5), add=T)
>hist(c, col="green", breaks=seq(-5,40,2.5), add=T)

やはり重ね合わせはうまくいかない。
特に6つのグラフが重なるような状況になると悲惨である。
>hist(a, col="blue",ylim=c(1,40),breaks=seq(-15,40,2.5))
>hist(b, col="red", breaks=seq(-15,40,2.5), add=T)
>hist(c, col="green", breaks=seq(-15,40,2.5), add=T)
>hist(d, col="orange", breaks=seq(-15,40,2.5), add=T)
>hist(e, col="yellow", breaks=seq(-15,40,2.5), add=T)
>hist(f, col="purple", breaks=seq(-15,40,2.5), add=T)

とまあひどい感じの重ね合わせになる。
一応biostaticsのページにreshape2とggplot2をつかってやる綺麗にhistgramを重ね合わせる方法がでているのであるが、このページと同じ内容をおこなってもmelt()がうまく働いてくれない(*)
(*)なんとなくバグっぽい!!!