今さらな話題で、今まで結構持ち上げておいてこの題名なの?となるところですが、ちょっと前にイスラエルハイテクベンチャーCEOの方が「アンチ・小保方STAP細胞の匿名ブログ『kahoの日記:STAP細胞の非実在について』のエセ科学性」というショッキングな題名でブログを書かれて話題になっていました。内容的にはいささか残念なところもあったので(*)、それに対抗するわけではありませんが、自分がハイテクベンチャーCEOだったら書いていただろう、kahoさんの日記への疑問点をいくつか述べてみようかなと思います。
本題に入る前に、Kahoさんの分析の一番の功績は前にもちょっと書いたように、Chip-seqのデーターにつかわれている細胞の由来が遺伝的バックグラウンドのことなるマウス由来であることを指摘されている点だと思います(**)。
ただこのことはデーターをdepositしてあるNCBIのサイトの情報をみれば以下のように、すぐわかることでした。
CD45+細胞:Oct3/4::gfp C57BL/6
ES細胞:C57BL/6 x 129/sv
STAP細胞:C57BL/6 x 129/sv
STAP幹細胞:C57BL/6 x 129/sv
ただふつう論文をさっと読むと、前提としてマウスの遺伝的バックグラウンドはそろっているものとして読むので、気が付きにくい点です。この部分をあえて分析して、多くの人にその事実を気づかせたということが一番大きいと思います。
こういう一部だけソースの違う細胞がつかってあるとか、Figごとに異なる細胞が使われてい場合、データーの出し方としてはunfairだなという感じがします。論文をまとめるうえで致し方ない状況があったのかもしれませんが、実験やっている人だとこういう形式の論文を見ると再現するのに苦労する結果(***)なのではないかなとうすうす感じます。なのでこの事実がわかった時点でもともとあった信頼性が半分以下になった感じですね。
Kahoさんのそれ以外の分析、TCRの再構成や細胞の由来の問題については、半分になった信頼がさらにどこまでおちるかというところにかかわっているだけで、個人的にはそれほど意義ある問題ではないと思っています。
そういったことを前提として、kahoさんのデーターをちょっといじってkahoさんの主張1)TCRの再構成、2)STAP細胞=ES細胞に対する疑問点を述べてみたいと思います。
1)TCRの再構成は本当にないいってよいのか?
これは前のブログのものづくりのための研究ノート026:STAP細胞にBCR再構成もないのか?のところでも同様の疑問が浮かんだのですが、
例えばSTAP細胞にTCRの再構成がないことを示すデーターの一つであるTCRbeta鎖の情報をみてみましょう。
TCR-beta
http://genome.ucsc.edu/cgi-bin/hgTracks?hgS_doOtherUser=submit&hgS_otherUserName=stopstap&hgS_otherUserSessionName=TCR%20beta%20rearrangement%20test
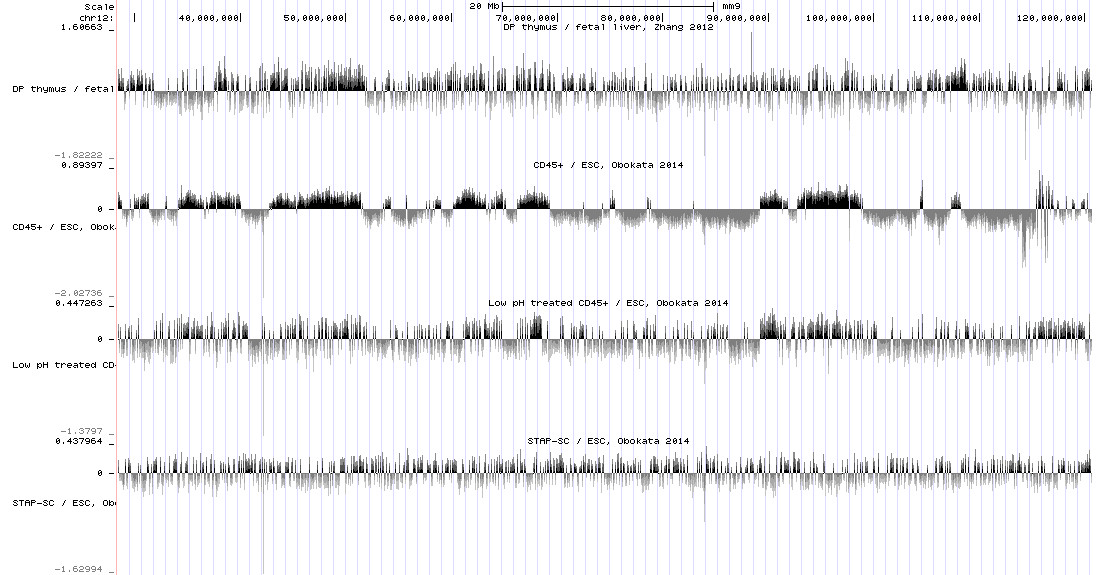
をみると次のような図1が出てきます。

この図はTCRベータ鎖遺伝子の部位のChip-seq input DNAのリードを、ポジコンのDPT細胞対FetalLiver(FL)細胞(1番目)、CD45対ES細胞(2番目)、STAP細胞(Low pH treated CD45+細胞)対ES細胞(3番目)、STAP幹細胞対ES細胞(4番目)で比較した図になります。この図からはこの部位のCD45+細胞のリード数がES細胞と比べて極端に少ないこと、またポジコンであるT細胞(DPT細胞)は血液幹細胞(FL)細胞と比べてリード数が同様に少なくなっていること、STAP細胞やSTAP幹細胞に関してはES細胞と比較してそのまでのリード数の差がないことが読み取れます。
このことから、STAP細胞やSTAP幹細胞ではTCR遺伝子の再構成がないと考えられるというのが、kahoさんの主張です。
ただこのCD45+/ES細胞で見られたリード数の差は本当にTCR遺伝子の再構成だけの影響なのかということは、きちんと検証しなければいけません。そうです恐ろしいことにこの二つの細胞は由来するマウスが違うためです。
このTCRベータ―鎖がある6番染色体全体をみてみましょう(上のUCSCサイトでZoom outしてみましょう)
すると次のような図2が現れます。

これをみるとCD45+/ES細胞の間のリード数が極端に異なる部位が、いくつもあることがわかるでしょう。つまりTCRと関係のない部位でもリード数が同程度異なる部分がいくつもあるのです。おそらくこれは、マウスの由来の違いでしょう。もしかするとTCRベータ鎖のところで認められたリード数の差もTCR遺伝子の再構成によるものでなく、マウスの由来の違いによるものかもしれません。またSTAP細胞とSTAP幹細胞はこの6番染色体のリード数のパターンをみるとちょっと違う細胞なので、もしかするとfairなコントロールをとると、STAP細胞にはTCR遺伝子の再構成が認められるという結果が得られるかもしれません。
2)STAP細胞=ES細胞なのか?
もう一つのKahoさんの分析の面白い点は、CNV(コピーナンバーバリエーション)という指標を用いて、CD45+細胞、STAP細胞、STAP幹細胞、ES細胞、TS細胞、FI幹細胞という細胞間の距離を導き出し、以下のように距離が一桁という少ない数字のため、ES細胞=STAP細胞=STAP幹細胞という結論をだされていることです。
ESC STAP-SC STAP FI-SC TSC
CD45+ 245 270 277 182 669
TSC 420 459 360 371
FI-SC 17 6 17
STAP 0 2
STAP-SC 6
ESC
ただこれも不思議なのは、図2のような染色体全体のリード数のパターンをみていると、CD45+細胞とES細胞は極端に違うこと(2番目)、またES細胞とSTAP幹細胞(4番目)は同様のパターンであることはわかるのですが、STAP細胞(3番目)に関してはCD45+細胞とES細胞もしくはSTAP幹細胞細胞の真ん中(というか足して2で割った)のような特徴があります。図2でも目で見てわかる程度にパターンが違います。またこの傾向はほかの染色体でも同様です。ここまで違うのに、CNVだと差がなくてよいのか?
見にくいのでここにも図2入れときます

つまりChip-seq input DNAのオーバーオールのリード数のSTAP細胞=ES細胞というCNVの結果と相関がなく、どちらが信頼性のある議論なのかという疑問がわきます。
(たしかに図2で見られるようなリード数だけの議論はDepthが少ない場合危険なのかもしれないけれど。。素人なんでこれ以上の議論は無理ですね)
もちろんここでの議論はUCSCにkahoさんがあげられたデーターをもとにしており、その分析やUCSCサイトで利用できるデーターが不十分でないことによるのかもしれません。またChip-seqのinputDNAのデータからいろいろなことがわかる可能性について指摘された功績も大きいと思います。ただ以上に挙げた理由から、上記2点についてはいささか言い過ぎのところもあるのではないかなと個人的には思っています(****)。
どこまで信じれるのか?については、答えになっていませんが、kahoさんのサイトのある人のコメントにもありましたが、「ここから先はやはり各々で解析して確かめるのが美しいスタンスでしょうね。」
私もちょっと勉強がてらいくつかのデータを分析しております。マシンのパワーがいまいちなので、なかなかタイムリーにはいきませんが、何か面白いことがわかればレポートしてみたいですね。こうご期待。
(*)ベンチャーのプロモーションのためなのかなと思いきや、重箱の隅系なcriticismでちょっと残念でした。せっかくハイテクベンチャーなのだから、kahoの日記への単なるcriticismだけでなくご自身の独自の分析をブログでだされるとビジネスにつながったのではないかな。ただ知名度を上げるには寄与したと思うので、50点かな。辛口かもしれませんが。。
(**)よく考えるとデーターをダウンロードするときに、kahoさんはマウスの由来が違うことに気付いたと思うのだけれど、確信犯で解析したのかしら?確かに、単にデータベースの記載から遺伝的バックグラウンドの違うマウスを解析しているというよりは、解析してもったいぶって情報を出した方が注目は集まると思うし、現に注目が集まった。
(***)あくまでも一般論ですが、チャンピオンデーターなどのケースが多い気がします。こういうデーターを見たら、よっぽどの理由(レビューワーからやってみろと言われたなど)がない限り、触らないのが無難なのですが、STAPはインパクトがありすぎたせいか触り続けています。吉村先生もなかなかやめられなかったからな。そろそろやめにしないと(笑)。
最近ものづくりのための研究ノートがこればかり。。本来はコアな技術論とかを書いてみたいのだけれど。。
(****)ここでの議論もkahoさんがUCSCに挙げられたデーターをもとに行いました。あしからず。
(追記)2016年3月1日追記。
個人的にはもはや興味を失っていたのだけれど、未だにこのブログのSTAP関連の項目を目にされている方が多い様なので、追記しておく。
2015年9月24日同様の趣旨からなる論文をNatureが掲載した。
1報目"Failure to replicate the STAP cell phenomenon"はボストンを中心とした幹細胞分野の大御所たちのチームによるもの
2報目"STAP cells are derived from ES cells"は理研のグループによるもの
の二つで、1はほぼ全てのSTAPのprotocolを検証し、作成できないことを示すとともに、バイオインフォマテッィクスを利用してSTAP細胞とされていたものに、ESが混ざっていることを証明したもの、2もバイオインフォマティックスを用いてESのまざりを証明したものである。
STAPの論文は途中から基本的に無理筋な感じを呈してきたが、これで完全に決着がついた。残念ながら、おそらく誰かがES細胞を混ぜて虚構の細胞を作ったことになる(結局それが誰かわからないのであるが。。)。ただこの一件で日本のサイエンスに大きな傷跡が残り、一人の偉大な科学者を失ったことは悔やまれる。
本題に入る前に、Kahoさんの分析の一番の功績は前にもちょっと書いたように、Chip-seqのデーターにつかわれている細胞の由来が遺伝的バックグラウンドのことなるマウス由来であることを指摘されている点だと思います(**)。
ただこのことはデーターをdepositしてあるNCBIのサイトの情報をみれば以下のように、すぐわかることでした。
CD45+細胞:Oct3/4::gfp C57BL/6
ES細胞:C57BL/6 x 129/sv
STAP細胞:C57BL/6 x 129/sv
STAP幹細胞:C57BL/6 x 129/sv
ただふつう論文をさっと読むと、前提としてマウスの遺伝的バックグラウンドはそろっているものとして読むので、気が付きにくい点です。この部分をあえて分析して、多くの人にその事実を気づかせたということが一番大きいと思います。
こういう一部だけソースの違う細胞がつかってあるとか、Figごとに異なる細胞が使われてい場合、データーの出し方としてはunfairだなという感じがします。論文をまとめるうえで致し方ない状況があったのかもしれませんが、実験やっている人だとこういう形式の論文を見ると再現するのに苦労する結果(***)なのではないかなとうすうす感じます。なのでこの事実がわかった時点でもともとあった信頼性が半分以下になった感じですね。
Kahoさんのそれ以外の分析、TCRの再構成や細胞の由来の問題については、半分になった信頼がさらにどこまでおちるかというところにかかわっているだけで、個人的にはそれほど意義ある問題ではないと思っています。
そういったことを前提として、kahoさんのデーターをちょっといじってkahoさんの主張1)TCRの再構成、2)STAP細胞=ES細胞に対する疑問点を述べてみたいと思います。
1)TCRの再構成は本当にないいってよいのか?
これは前のブログのものづくりのための研究ノート026:STAP細胞にBCR再構成もないのか?のところでも同様の疑問が浮かんだのですが、
例えばSTAP細胞にTCRの再構成がないことを示すデーターの一つであるTCRbeta鎖の情報をみてみましょう。
TCR-beta
http://genome.ucsc.edu/cgi-bin/hgTracks?hgS_doOtherUser=submit&hgS_otherUserName=stopstap&hgS_otherUserSessionName=TCR%20beta%20rearrangement%20test
をみると次のような図1が出てきます。

この図はTCRベータ鎖遺伝子の部位のChip-seq input DNAのリードを、ポジコンのDPT細胞対FetalLiver(FL)細胞(1番目)、CD45対ES細胞(2番目)、STAP細胞(Low pH treated CD45+細胞)対ES細胞(3番目)、STAP幹細胞対ES細胞(4番目)で比較した図になります。この図からはこの部位のCD45+細胞のリード数がES細胞と比べて極端に少ないこと、またポジコンであるT細胞(DPT細胞)は血液幹細胞(FL)細胞と比べてリード数が同様に少なくなっていること、STAP細胞やSTAP幹細胞に関してはES細胞と比較してそのまでのリード数の差がないことが読み取れます。
このことから、STAP細胞やSTAP幹細胞ではTCR遺伝子の再構成がないと考えられるというのが、kahoさんの主張です。
ただこのCD45+/ES細胞で見られたリード数の差は本当にTCR遺伝子の再構成だけの影響なのかということは、きちんと検証しなければいけません。そうです恐ろしいことにこの二つの細胞は由来するマウスが違うためです。
このTCRベータ―鎖がある6番染色体全体をみてみましょう(上のUCSCサイトでZoom outしてみましょう)
すると次のような図2が現れます。

これをみるとCD45+/ES細胞の間のリード数が極端に異なる部位が、いくつもあることがわかるでしょう。つまりTCRと関係のない部位でもリード数が同程度異なる部分がいくつもあるのです。おそらくこれは、マウスの由来の違いでしょう。もしかするとTCRベータ鎖のところで認められたリード数の差もTCR遺伝子の再構成によるものでなく、マウスの由来の違いによるものかもしれません。またSTAP細胞とSTAP幹細胞はこの6番染色体のリード数のパターンをみるとちょっと違う細胞なので、もしかするとfairなコントロールをとると、STAP細胞にはTCR遺伝子の再構成が認められるという結果が得られるかもしれません。
2)STAP細胞=ES細胞なのか?
もう一つのKahoさんの分析の面白い点は、CNV(コピーナンバーバリエーション)という指標を用いて、CD45+細胞、STAP細胞、STAP幹細胞、ES細胞、TS細胞、FI幹細胞という細胞間の距離を導き出し、以下のように距離が一桁という少ない数字のため、ES細胞=STAP細胞=STAP幹細胞という結論をだされていることです。
ESC STAP-SC STAP FI-SC TSC
CD45+ 245 270 277 182 669
TSC 420 459 360 371
FI-SC 17 6 17
STAP 0 2
STAP-SC 6
ESC
ただこれも不思議なのは、図2のような染色体全体のリード数のパターンをみていると、CD45+細胞とES細胞は極端に違うこと(2番目)、またES細胞とSTAP幹細胞(4番目)は同様のパターンであることはわかるのですが、STAP細胞(3番目)に関してはCD45+細胞とES細胞もしくはSTAP幹細胞細胞の真ん中(というか足して2で割った)のような特徴があります。図2でも目で見てわかる程度にパターンが違います。またこの傾向はほかの染色体でも同様です。ここまで違うのに、CNVだと差がなくてよいのか?
見にくいのでここにも図2入れときます

つまりChip-seq input DNAのオーバーオールのリード数のSTAP細胞=ES細胞というCNVの結果と相関がなく、どちらが信頼性のある議論なのかという疑問がわきます。
(たしかに図2で見られるようなリード数だけの議論はDepthが少ない場合危険なのかもしれないけれど。。素人なんでこれ以上の議論は無理ですね)
もちろんここでの議論はUCSCにkahoさんがあげられたデーターをもとにしており、その分析やUCSCサイトで利用できるデーターが不十分でないことによるのかもしれません。またChip-seqのinputDNAのデータからいろいろなことがわかる可能性について指摘された功績も大きいと思います。ただ以上に挙げた理由から、上記2点についてはいささか言い過ぎのところもあるのではないかなと個人的には思っています(****)。
どこまで信じれるのか?については、答えになっていませんが、kahoさんのサイトのある人のコメントにもありましたが、「ここから先はやはり各々で解析して確かめるのが美しいスタンスでしょうね。」
私もちょっと勉強がてらいくつかのデータを分析しております。マシンのパワーがいまいちなので、なかなかタイムリーにはいきませんが、何か面白いことがわかればレポートしてみたいですね。こうご期待。
(*)ベンチャーのプロモーションのためなのかなと思いきや、重箱の隅系なcriticismでちょっと残念でした。せっかくハイテクベンチャーなのだから、kahoの日記への単なるcriticismだけでなくご自身の独自の分析をブログでだされるとビジネスにつながったのではないかな。ただ知名度を上げるには寄与したと思うので、50点かな。辛口かもしれませんが。。
(**)よく考えるとデーターをダウンロードするときに、kahoさんはマウスの由来が違うことに気付いたと思うのだけれど、確信犯で解析したのかしら?確かに、単にデータベースの記載から遺伝的バックグラウンドの違うマウスを解析しているというよりは、解析してもったいぶって情報を出した方が注目は集まると思うし、現に注目が集まった。
(***)あくまでも一般論ですが、チャンピオンデーターなどのケースが多い気がします。こういうデーターを見たら、よっぽどの理由(レビューワーからやってみろと言われたなど)がない限り、触らないのが無難なのですが、STAPはインパクトがありすぎたせいか触り続けています。吉村先生もなかなかやめられなかったからな。そろそろやめにしないと(笑)。
最近ものづくりのための研究ノートがこればかり。。本来はコアな技術論とかを書いてみたいのだけれど。。
(****)ここでの議論もkahoさんがUCSCに挙げられたデーターをもとに行いました。あしからず。
(追記)2016年3月1日追記。
個人的にはもはや興味を失っていたのだけれど、未だにこのブログのSTAP関連の項目を目にされている方が多い様なので、追記しておく。
2015年9月24日同様の趣旨からなる論文をNatureが掲載した。
1報目"Failure to replicate the STAP cell phenomenon"はボストンを中心とした幹細胞分野の大御所たちのチームによるもの
2報目"STAP cells are derived from ES cells"は理研のグループによるもの
の二つで、1はほぼ全てのSTAPのprotocolを検証し、作成できないことを示すとともに、バイオインフォマテッィクスを利用してSTAP細胞とされていたものに、ESが混ざっていることを証明したもの、2もバイオインフォマティックスを用いてESのまざりを証明したものである。
STAPの論文は途中から基本的に無理筋な感じを呈してきたが、これで完全に決着がついた。残念ながら、おそらく誰かがES細胞を混ぜて虚構の細胞を作ったことになる(結局それが誰かわからないのであるが。。)。ただこの一件で日本のサイエンスに大きな傷跡が残り、一人の偉大な科学者を失ったことは悔やまれる。