まずRでベン図を描いてみる (Making Venn diagrams)
データベースにディポジットされている各種遺伝子変異とそれに関連してCpGがHypermethylationされている遺伝子のリストDNAmet.csvをexcelで作成。
その後は
> x <- read.table(file="DNAmet.csv",header=T,sep=",")
で読み込み
>head(x)
# a b c d e
#1 PYROXD2 HPSE2 LOXL4 ABCC2 KAZALD1
#2 ENTPD7 ENTPD7 HPSE2 SH3PXD2A BTRC
#3 BLOC1S2 SCD ENTPD7 VWA2 FBXW4
#4 FAM178A FGF8 ABCC2 ADARB2 NOLC1
#5 FBXW4 ABLIM1 SCD CHST15 ELOVL3
#6 KCNIP2 ECHDC3 NDUFB8 DOCK1 SH3PXD2A
こんな感じのファイルである(カラム名のa,b,cは各遺伝子変異に相当)。要素の遺伝子数がそれぞれ異なるので、変異遺伝子ごとに遺伝子リストマトリックスを作成する。
> a <- as.matrix(x[,1])
> b <- as.matrix(x[,2][1:1135])
> c <- as.matrix(x[,3][1:4064])
> d <- as.matrix(x[,4][1:761])
> e <- as.matrix(x[,5][1:2450])
a,b,c遺伝子変異の間で共通するメチル化遺伝子のベン図をかかせるためには、
これをリスト型のオブジェクトに変え
> data <- list(a,b,c)
gplotsのvenn関数を利用すれば簡単にベン図が書ける
> library(gplots)
> venn(data)
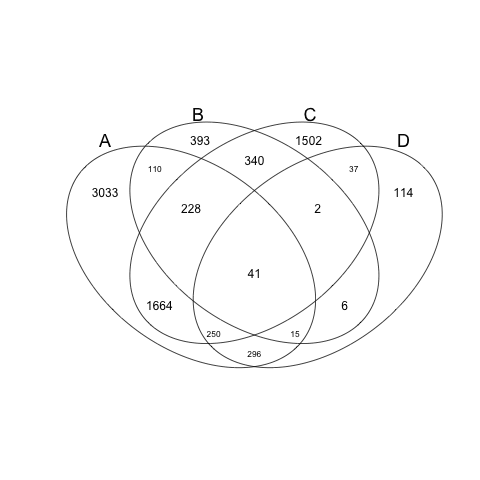
4つ以上のベン図もVennでかける(making venn diagram with more than 4 elements)
同様に4つのベン図もVennでかける
> data2 <- list(a,b,c,d)
> venn(data2)
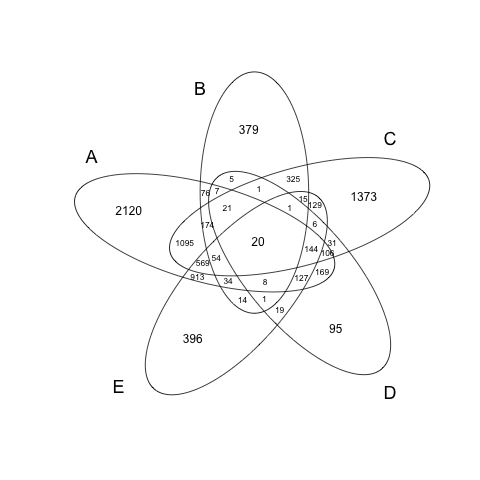
同様に5つのベン図もOK
> data3 <- list(a,b,c,d,e)
> venn(data3)

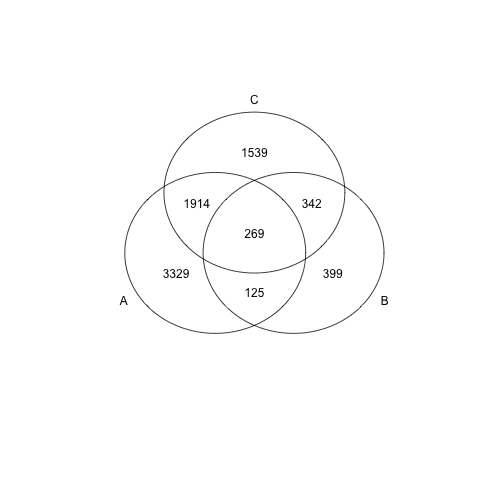
Venn図のデーター要素をとるためには? (How to make a gene list from a venn diagram)
例えばこの図の真ん中の269に含まれるgene nameを出したい場合は、

intersect関数をつかって、a&b&cをt1に入れるといい。
> t1 <- intersect(a,intersect(b,c))
> t1 <- as.matrix(t1)
t1の列数(に含まれる遺伝子数)を見ると
>nrow(t1)
[1] 269
となっている。
実際には
>head(t1)
[1] "ENTPD7" "FAM53B" "DOCK1" "FAM196A" "EBF3" "BNIP3"
といった遺伝子が含まれている。
あとは
>write.table(t1,file="overlap.txt")
でテキストファイルに書き出せばエエやろ!
データベースにディポジットされている各種遺伝子変異とそれに関連してCpGがHypermethylationされている遺伝子のリストDNAmet.csvをexcelで作成。
その後は
> x <- read.table(file="DNAmet.csv",header=T,sep=",")
で読み込み
>head(x)
# a b c d e
#1 PYROXD2 HPSE2 LOXL4 ABCC2 KAZALD1
#2 ENTPD7 ENTPD7 HPSE2 SH3PXD2A BTRC
#3 BLOC1S2 SCD ENTPD7 VWA2 FBXW4
#4 FAM178A FGF8 ABCC2 ADARB2 NOLC1
#5 FBXW4 ABLIM1 SCD CHST15 ELOVL3
#6 KCNIP2 ECHDC3 NDUFB8 DOCK1 SH3PXD2A
こんな感じのファイルである(カラム名のa,b,cは各遺伝子変異に相当)。要素の遺伝子数がそれぞれ異なるので、変異遺伝子ごとに遺伝子リストマトリックスを作成する。
> a <- as.matrix(x[,1])
> b <- as.matrix(x[,2][1:1135])
> c <- as.matrix(x[,3][1:4064])
> d <- as.matrix(x[,4][1:761])
> e <- as.matrix(x[,5][1:2450])
a,b,c遺伝子変異の間で共通するメチル化遺伝子のベン図をかかせるためには、
これをリスト型のオブジェクトに変え
> data <- list(a,b,c)
gplotsのvenn関数を利用すれば簡単にベン図が書ける
> library(gplots)
> venn(data)

4つ以上のベン図もVennでかける(making venn diagram with more than 4 elements)
同様に4つのベン図もVennでかける
> data2 <- list(a,b,c,d)
> venn(data2)

同様に5つのベン図もOK
> data3 <- list(a,b,c,d,e)
> venn(data3)

Venn図のデーター要素をとるためには? (How to make a gene list from a venn diagram)
例えばこの図の真ん中の269に含まれるgene nameを出したい場合は、

intersect関数をつかって、a&b&cをt1に入れるといい。
> t1 <- intersect(a,intersect(b,c))
> t1 <- as.matrix(t1)
t1の列数(に含まれる遺伝子数)を見ると
>nrow(t1)
[1] 269
となっている。
実際には
>head(t1)
[1] "ENTPD7" "FAM53B" "DOCK1" "FAM196A" "EBF3" "BNIP3"
といった遺伝子が含まれている。
あとは
>write.table(t1,file="overlap.txt")
でテキストファイルに書き出せばエエやろ!