次にやりたいのは、boxplotとdotplotの重ね合わせでしょうか?
#乱数で、サンプル数100, 平均20, 標準偏差5のデーターをつくる
> a<-rnorm(100,20,5)
#乱数で、サンプル数60, 平均10, 標準偏差3のデーターをつくる
> b<-rnorm(60,10,3)
#乱数で、サンプル数80 平均5, 標準偏差3のデーターをつくる
> c<-rnorm(80,5,3)
ここで問題となるのは、dotplotを生成する関数stripchart()が複数のオブジェクトを入れられないことです。
そこでdata.frame()をつかってデーターフレームを作りたいところですが、このケースのようにサンプル数が違う場合data.frameが働きません。そこでb, cのあまりの部分に空の値である。NAを放り込んで、サンプル数を同じにしてデーターフレームをつくってみます。
#b[61]~b[100]にNAを入れる
> b[61:100]<-NA
#c[81]~c[100]にNAを入れる
> c[81:100] <-NA
#ベクトルa,b,cからなるデーターフレームを作る
> data<- data.frame(a,b,c)
#確認
> data
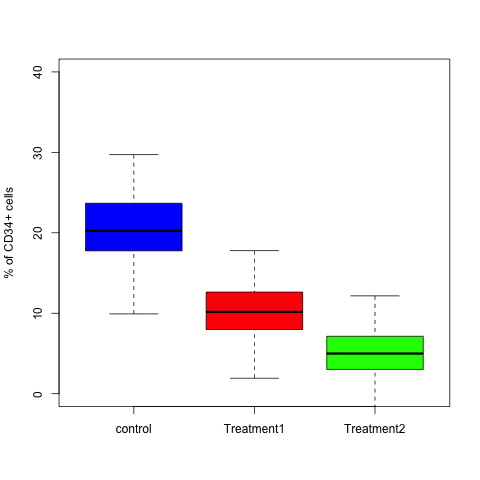
ここまでくればだいたいOKでしょう。

といったグラフが得られるでしょう。
このままだとやはりみにくいので、
> boxplot(data, ylim=c(0,35),outline=F,col=c("Blue", "Red", "Green"),names=c("control","Treatment1", "Treatment2"),plot=T,ylab="% of CD34+ cells")
> stripchart(data, vertical = TRUE, pch = 21, col = "black", bg =NA,method = "jitter", add = TRUE)
として

とか
> boxplot(data, ylim=c(0,35),outline=F,col=c("Blue", "Red", "Green"),names=c("control","Treatment1", "Treatment2"),plot=T,ylab="% of CD34+ cells")
> stripchart(data, vertical = TRUE, pch = 21, col = "maroon", bg ="orange",method = "jitter", add = TRUE)
として
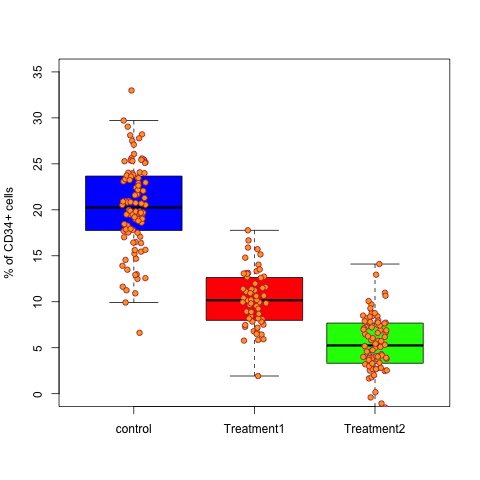
とかグラフをつくってみるとよいのではないでしょうか?
*Rスクリプトをうまくブログに乗せる方法がわからず、以前の表記に戻しました。
#乱数で、サンプル数100, 平均20, 標準偏差5のデーターをつくる
> a<-rnorm(100,20,5)
#乱数で、サンプル数60, 平均10, 標準偏差3のデーターをつくる
> b<-rnorm(60,10,3)
#乱数で、サンプル数80 平均5, 標準偏差3のデーターをつくる
> c<-rnorm(80,5,3)
ここで問題となるのは、dotplotを生成する関数stripchart()が複数のオブジェクトを入れられないことです。
そこでdata.frame()をつかってデーターフレームを作りたいところですが、このケースのようにサンプル数が違う場合data.frameが働きません。そこでb, cのあまりの部分に空の値である。NAを放り込んで、サンプル数を同じにしてデーターフレームをつくってみます。
#b[61]~b[100]にNAを入れる
> b[61:100]<-NA
#c[81]~c[100]にNAを入れる
> c[81:100] <-NA
#ベクトルa,b,cからなるデーターフレームを作る
> data<- data.frame(a,b,c)
#確認
> data
ここまでくればだいたいOKでしょう。

といったグラフが得られるでしょう。
このままだとやはりみにくいので、
> boxplot(data, ylim=c(0,35),outline=F,col=c("Blue", "Red", "Green"),names=c("control","Treatment1", "Treatment2"),plot=T,ylab="% of CD34+ cells")
> stripchart(data, vertical = TRUE, pch = 21, col = "black", bg =NA,method = "jitter", add = TRUE)
として

とか
> boxplot(data, ylim=c(0,35),outline=F,col=c("Blue", "Red", "Green"),names=c("control","Treatment1", "Treatment2"),plot=T,ylab="% of CD34+ cells")
> stripchart(data, vertical = TRUE, pch = 21, col = "maroon", bg ="orange",method = "jitter", add = TRUE)
として

とかグラフをつくってみるとよいのではないでしょうか?
*Rスクリプトをうまくブログに乗せる方法がわからず、以前の表記に戻しました。