ご訪問ありがとうございます。 昨日までの状況は右下の「アクセス状況」にあります。
せっかく、「MOPAC2009を導入」したので、PM6法の実力(?)を試してみました。
NaCl(塩化ナトリウム)の構造を作ってみました。
(Na、Cl 原子各4ヶから成るもの)
よく例にする、CH3COOH などは、共有結合している、分子ですね。
(共有結合した原子団を分子と云う)
でも、NaCl などは、イオン結合している唯の原子団?ですね。
さて、
モデリングソフトは当然ながら「Winmostar」です。
(1)最初に1分子分を構造最適化する。(1次元)
「Winmostar」立ち上げ時に表れる、C-H 構造の C を Na に変え、H を Cl に変え、
「MOPAC2009」のパラメータは「PM3 EF PRECISE」とする(此処は、PM3法です)。
計算実行=構造最適化。

(2)2分子を配置する。(2次元化)
まず、適当に、Na を追加する。(Cl 側でなく、あえて Na 側にする)
(Na 同士が結合しない程度に離して)
次に、Cl を追加する。(追加した Na から少し離して、図のような位置に)
(追加した、Na と Cl は自動的に結合される)

(3)2分子分を構造最適化する。(事前最適化)
「MOPAC2009」のパラメータは同じく「PM3 EF PRECISE」とする(一旦は、PM3法で)。
計算実行=構造最適化。

(4)再度、2分子分を構造最適化する。(2次元=平面 完成)
「MOPAC2009」のパラメータは今度は「PM6 EF PRECISE」とする(本番)。
計算実行=構造最適化。

(5)4分子を配置する。(3次元化)
まず、「Winmostar」の「編集」メニューから「鏡像体生成」を行う。
少し離れすぎている?ようなので、「編集」メニューから「部分移動」を選んで、
2組の2分子間を少し近づける。
(面が平行でないが、この方が分かりやすい?)

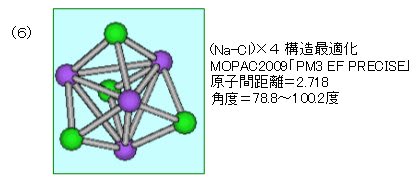
(6)4分子分を構造最適化する。(事前最適化)
「MOPAC2009」のパラメータは「PM3 EF PRECISE」とする(一旦は、PM3法で)。
計算実行=構造最適化。
(7)再度、4分子分を構造最適化する。(3次元=立体 完成)
「MOPAC2009」のパラメータは今度は「PM6 EF PRECISE」とする(本番)。
計算実行=構造最適化。
結果は、原子間距離=2.887 で、角度は87.3/92.6度 となった。

正解(?)は ・・・・・
「研究所一般公開 見学」で書いた、「結晶構造ギャラリー」で確認。
原子間距離=2.820 で 角度=90.0度 の正六面体(立方体)ですね。
と云うことは、(半経験的分子軌道法計算での)誤差は2~3%と云うことですネ。
素晴らしい!!! これがPM6法の実力(?)ですね???
と云うことで、今日の 部活動 は終了です。
見ていただきありがとうございました。
お帰りに投票して頂けると嬉しいです。 ⇒ (「60代~」と「茨城」)
(「60代~」と「茨城」)
人気BlogRanking ⇒ (「インターネット・・・・・」>「ウェブログ」)
(「インターネット・・・・・」>「ウェブログ」)
P-NETBANKING ⇒ (「パソコン・・・・・」と「茨城」)
(「パソコン・・・・・」と「茨城」)
せっかく、「MOPAC2009を導入」したので、PM6法の実力(?)を試してみました。
NaCl(塩化ナトリウム)の構造を作ってみました。
(Na、Cl 原子各4ヶから成るもの)
よく例にする、CH3COOH などは、共有結合している、分子ですね。
(共有結合した原子団を分子と云う)
でも、NaCl などは、イオン結合している唯の原子団?ですね。
さて、
モデリングソフトは当然ながら「Winmostar」です。
(1)最初に1分子分を構造最適化する。(1次元)
「Winmostar」立ち上げ時に表れる、C-H 構造の C を Na に変え、H を Cl に変え、
「MOPAC2009」のパラメータは「PM3 EF PRECISE」とする(此処は、PM3法です)。
計算実行=構造最適化。

(2)2分子を配置する。(2次元化)
まず、適当に、Na を追加する。(Cl 側でなく、あえて Na 側にする)
(Na 同士が結合しない程度に離して)
次に、Cl を追加する。(追加した Na から少し離して、図のような位置に)
(追加した、Na と Cl は自動的に結合される)

(3)2分子分を構造最適化する。(事前最適化)
「MOPAC2009」のパラメータは同じく「PM3 EF PRECISE」とする(一旦は、PM3法で)。
計算実行=構造最適化。

(4)再度、2分子分を構造最適化する。(2次元=平面 完成)
「MOPAC2009」のパラメータは今度は「PM6 EF PRECISE」とする(本番)。
計算実行=構造最適化。

(5)4分子を配置する。(3次元化)
まず、「Winmostar」の「編集」メニューから「鏡像体生成」を行う。
少し離れすぎている?ようなので、「編集」メニューから「部分移動」を選んで、
2組の2分子間を少し近づける。
(面が平行でないが、この方が分かりやすい?)

(6)4分子分を構造最適化する。(事前最適化)
「MOPAC2009」のパラメータは「PM3 EF PRECISE」とする(一旦は、PM3法で)。
計算実行=構造最適化。

(7)再度、4分子分を構造最適化する。(3次元=立体 完成)
「MOPAC2009」のパラメータは今度は「PM6 EF PRECISE」とする(本番)。
計算実行=構造最適化。
結果は、原子間距離=2.887 で、角度は87.3/92.6度 となった。

正解(?)は ・・・・・
「研究所一般公開 見学」で書いた、「結晶構造ギャラリー」で確認。
原子間距離=2.820 で 角度=90.0度 の正六面体(立方体)ですね。
と云うことは、(半経験的分子軌道法計算での)誤差は2~3%と云うことですネ。
素晴らしい!!! これがPM6法の実力(?)ですね???
と云うことで、今日の 部活動 は終了です。

見ていただきありがとうございました。
お帰りに投票して頂けると嬉しいです。 ⇒
 (「60代~」と「茨城」)
(「60代~」と「茨城」)人気BlogRanking ⇒
 (「インターネット・・・・・」>「ウェブログ」)
(「インターネット・・・・・」>「ウェブログ」)P-NETBANKING ⇒
 (「パソコン・・・・・」と「茨城」)
(「パソコン・・・・・」と「茨城」)








